Dziewięciu wspaniałych cz.I
W ciągu ostatnich lat komputer stał się podstawowym narzędziem umożliwiającym projektowanie leków. Proces ten można prowadzić z użyciem różnych metodologii – każda z nich ma swoje plusy i minusy. Które z nich warto wybrać, jak się nimi posługiwać i jakich rezultatów się spodziewać? Przedstawiamy dziewięć najpopularniejszych.
Rozwój medycyny i farmacji w XX wieku przyczynił się do znacznego wydłużenia czasu i jakości życia. Nadal jest jednak wiele chorób, które przysparzają pacjentom cierpień, a na które nie mamy dobrego lekarstwa. Zdarza się, że leki znajdujące się w naszej dyspozycji mają wiele uciążliwych działań niepożądanych albo są skuteczne tylko do pewnego czasu, jak w przypadku wielu nowotworów. Tak więc, pomimo znalezienia kilku tysięcy substancji leczniczych, cały czas polujemy na nowe leki.
.jpg) |
| Rys. 1. Metody komputerowego projektowania leków przeżywają gwałtowny rozwój, co ilustruje dynamiczny wzrost liczby publikacji dotyczących CADD przez ostatnich 15 lat. |
Projektowanie leków
Dawniej przypominało to szukanie igły w stogu siana, a sukces wynikał nierzadko ze szczęśliwego przypadku Jednak wraz z rozwojem podstaw teoretycznych opisu
mechanizmów działania leków (głównie za sprawą osiągnięć chemii obliczeniowej i biologii strukturalnej) możliwe stało się bardziej racjonalne podejście do projektowania leków. Szybki wzrost mocy obliczeniowej komputerów ułatwił wykorzystanie tej wiedzy w specjalistycznych programach komputerowych, które znacznie usprawniają proces badawczy. I tak oto w ciągu ostatnich 10-15 lat komputer przestał być w laboratoriach sprzętem jedynie do przechowywania i opisywania wyników czy też prowadzenia statystyk, a stał się podstawowym narzędziem umożliwiającym projektowanie leków (rys. 1).
Burzliwy rozwój komputerowo-wspomaganego projektowania leków (ang. computer-aided drug design, CADD) w ostatnich latach zaowocował powstaniem wielu metodologii mających pomóc w pracach nad lekiem. Spośród nich krótko opisanych zostanie dziewięć najczęściej używanych (tytułowych dziewięciu wspaniałych).
Podglądanie leków i białek
Większość leków działa przez aktywowanie bądź blokowanie wybranych białek – receptorów i enzymów. Cząsteczka lecznicza łączy się z miejscem wiążącym białka i w większości przypadków indukuje niewielką zmianę w jego strukturze trójwymiarowej, co powoduje powstanie pewnej informacji biologicznej i przekazanie jej dalej przez kaskady sygnałowe, a następnie określony skutek na poziomie komórki, tkanki, narządu i całego organizmu.
Oddziaływanie leku z białkiem przypomina dopasowanie klucza i zamka. Cząsteczka aktywna musi mieć odpowiedni kształt przestrzenny, by zmieścić się w miej-scu wiążącym receptora. Co więcej, aby wiązanie to było trwałe, musi posiadać odpowiednio zlokalizowane grupy funkcyjne zdolne do tworzenia oddziaływań międzycząsteczkowych (np. wiązań wodorowych), które utrzymają lek w kieszeni wiążącej tak długo, aż możliwe będzie powstanie efektu biologicznego.
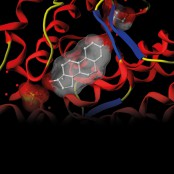
Jeśli dzięki krystalografii bądź spektroskopii NMR znamy kształt kompleksu białka z lekiem, to możliwe jest komputerowe „podglądanie” działania leku. Grafika komputerowa pozwala na łatwą do przyswojenia dla badacza i farmaceuty wizualizację oddziaływań poszczególnych grup funkcyjnych związku leczniczego z aminokwasami receptora (rys. 2).
.jpg) |
| Rys. 2. Grafika komputerowa umożliwia łatwą analizę kompleksów lek-białko. |
Analiza i wizualizacja tych oddziaływań oraz budowy miejsca wiążącego może podpowiadać potencjalne modyfikacje w strukturze związku, które wzmocnią oddziaływanie substancji z receptorem (rys. 3).
Dla większości związków nie jest jednak możliwe rozwiązanie struktur krystalograficznych ich kompleksów z białkami. Z pomocą przychodzi wtedy dokowanie molekularne, które wychodząc ze struktur miejsca wiążącego i leku, stara się przewidzieć, w jaki sposób łączą się one ze sobą oraz oszacować siłę takiego oddziaływania.
Programy dokujące rozwiązują zazwyczaj trzy problemy:
1. która z wielu możliwych konformacji ligandu jest konformacją bioaktywną? (niestety, nie zawsze jest to konformacja o najniższej energii); 2. jak wpasowuje się ona do miejsca wiążącego?; 3. jaka jest siła oddziaływania między ligandem i białkiem? Ponieważ przestrzeń rozwiązań problemów 1. i 2. jest niesłychanie szeroka i wielowymiarowa, trudnym zagadnieniem jest rozsądne jej zmniejszenie tak, by jednocześnie nie „zgubić” prawidłowego rozwiązania. Stanowi to spore wyzwanie, ale ponad 20 lat rozwoju algorytmów optymalizacyjnych doprowadziło do stanu, w którym na zwykłym komputerze PC można wygenerować prawidłową (lub bliską prawidłowej) geometrię przestrzenną kompleksu lek-białko w czasie poniżej minuty. Dużo gorzej radzimy sobie z trzecim problemem. Niestety, funkcje oceniające nadal nie potrafią zbyt dobrze szacować siły wiązania leku – rzadko w którym przypadku udaje się uzyskać korelację pomiędzy danymi eksperymentalnymi a przewidywaniami powyżej 40-50%, co nie jest wynikiem satysfakcjonującym i nie pozwala wykorzystać metodologii dokowania w takim stopniu, jakbyśmy tego chcieli.
.jpg) |
| Rys. 3. Zagadnienie dokowania sprowadza się do pytania: jak wygląda kompleks powstały z połączenia leku z miejscem wiążącym białka? |
Igła w stogu siana czy może coś zupełnie nowego?
Pomimo powyższych problemów dokowanie molekularne jest prawdopodobnie najpowszechniej stosowaną metodą komputerową w projektowaniu leków. Jednym z ważniejszych jej zastosowań jest przesiewanie wirtualne (ang. virtual screening, VS), stosowane często przy poszukiwaniu nowych związków wiodących.
Przesiewanie wirtualne jest komputerowym odpowiednikiem doświadczalnego przesiewania wielkoprzepustowego (ang. high-throughoutput screening, HTS). Przemysł farmaceutyczny pokładał wielkie nadzieje w tej technologii, która miała dzięki masowej skali przynieść ilościowy skok nowych leków. Eksperyment HTS polega na zautomatyzowanym mierzeniu powinowactwa setek bądź nawet tysięcy związków wobec danego celu molekularnego. Niestety, pomimo początkowego entuzjazmu, okazało się, że koszt generowany przez HTS wcale nie zwraca się w postaci dużej liczby nowych związków aktywnych.Podstawową przyczyną tego stanu rzeczy jest nieukierunkowane stosowanie HTS. Zasada „wrzućmy wszystko i zobaczmy, co się stanie” zwykle nie przynosi dobrych efektów. Dodatkowo, o wielu związkach od razu można powiedzieć, że nie będą aktywne bądź nie będą się nadawały na leki z racji za dużej wagi cząsteczkowej, toksyczności etc.
Przeprowadzono więc racjonalizację stosowania HTS, a niemały udział miał tutaj właśnie przesiew wirtualny, który za pomocą dokowania wskazuje związki warte dalszych badań doświadczalnych. Procedura VS pozwala zredukować liczbę badanych związków z wielu tysięcy do na przykład kilkudziesięciu, mimo tego jednocześnie zwiększając szansę znalezienia właściwego związku aktywnego. Komputer jednak nie tylko może za nas poszukiwać igły w stogu siana szybciej i taniej, ale również zaproponować coś nowego. W metodach de novo, polegających na wirtualnym konstruowaniu cząsteczki leku z „cegiełek” atomów, grup funkcyjnych i łańcuchów, również szeroko używane jest dokowanie (rys. 4).
Programy de novo posiadają wbudowaną bibliotekę takich cegiełek i dopasowują je do miejsca wiążącego białka. Jeśli któryś z fragmentów zostaje oceniony wysoko, jako korzystnie oddziałujący z białkiem, zostaje on „zakotwiczony” w wybranym miejscu, a w jego sąsiedztwie dokowane są inne fragmenty. W podobny sposób powstaje kilka innych kotwic, które w końcu zostają zespolone w jedną molekułę chemiczną za pomocą łączników.
Ważną zaletą podejścia de novo jest otrzymywanie najczęściej zupełnie nowych chemotypów. Niestety, nie wszystkie zaprojektowane w ten sposób związki mogą być w rzeczywistości zsyntetyzowane. Jak do tej pory żaden nowy lek wygenerowany z metod de novo nie przeszedł wszystkich prób klinicznych, niemniej jednak w literaturze można znaleźć wiele przykładów, gdy zaproponowana przez algorytm de novo cząsteczka stała się podstawowym związkiem wiodącym, podlegającym dalszej optymalizacji na drodze teoretycznej i doświadczalnej.
.jpg) |
| Rys. 4. W metodach de novo program komputerowy konstruuje cząsteczkę nowego leku z „cegiełek” molekularnych dokowanych do receptora. |
Modelowanie homologiczne
Powyższe metody (jak również wiele innych) opierają się na znajomości budowy trójwymiarowej białka oraz jego miejsca wiążącego. Jeśli takich danych brak, można
symulować strukturę receptora za pomocą modelowania homologicznego.
W biologii ewolucyjnej jako homologiczne określa się biomolekuły, które mają wspólne pochodzenie. Dawno już dostrzeżono, że spokrewnione ze sobą białka, pomimo często licznych zmian w sekwencji aminokwasowej, posiadają zasadniczo podobną budowę przestrzenną. Jeśli zatem znamy strukturę trójwymiarową pewnego białka, to możliwe jest znalezienie przybliżonej struktury dowolnego białka do niego homologicznego.
Proces ten przebiega w kilku krokach. Najpierw przeprowadza się uliniowienie sekwencji białka modelowanego i białka szablonu tak, aby odpowiadające sobie aminokwasy leżały w identycznych pozycjach. Potem następuje przepisanie współrzędnych atomów z szablonu na atomy badanego białka. Jeśli w strukturze naszego receptora mamy do czynienia z insercjami, które nie mają odpowiedników w szablonie, oddzielnym etapem procedury jest modelowanie właśnie tych elementów. Następnie optymalizowane są łańcuchy boczne aminokwasów, a także struktura całego modelu, która podlega ocenie statystycznej. Kroki te są powtarzane aż do uzyskania jak najdokładniejszego modelu trójwymiarowego. Bardzo dobrą praktyką jest wprowadzanie do modelowania homologicznego wyników eksperymentalnych, np. pochodzących z eksperymentów mutagenezy danych o oddziaływaniu ligandów z poszczególnymi aminokwasami białka.
Uzyskany model homologiczny może następnie posłużyć do dokowania i procedury przesiewu wirtualnego.
Autorzy: Piotr F.J. Lipiński, Instytut Chemii Przemysłowej im. prof. Ignacego Mościckiego, Pracownia Spektroskopii i Modelowania Molekularnego, Dariusz Plewczyński, Zakład Chemii Fizycznej, Warszawski Uniwersytet Medyczny, Wydział Farmaceutyczny Centrum Modelowania Matematycznego i Komputerowego, ICM, Uniwersytet Warszawski
Artykuł został opublikowany w magazynie "Przemysł Farmaceutyczny" nr 5/2012
Fot.: Dariusz Plewczyński, Piotr F.J. Lipiński




